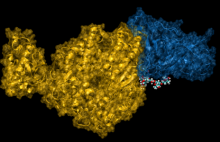
Des chercheurs du LPCT*, coordonnés par Antonio Monari, ont décidé d’utiliser la simulation moléculaire pour comprendre le comportement du Covid et proposer ainsi des médicaments rationnellement développés.
Simuler le microscopique pour comprendre le fonctionnement du virus
La lutte contre la pandémie de Covid-19 passe nécessairement par l’utilisation de la modélisation et de la simulation. Au-delà de la modélisation macroscopique du comportement des épidémies, dont nous sommes malgré nous devenus familiers, il est aussi possible d’utiliser des techniques numériques pour comprendre et visualiser quels sont les mécanismes microscopiques, à l’échelle des atomes, qui permettent le fonctionnement du virus et expliquent sa contagiosité et mortalité. Bien évidemment, comprendre ses mécanismes peut permettre d’identifier les médicaments potentiels qui pourront être envisagés pour freiner la contagion ou limiter les effets sur les organismes humaines. Il s’agit donc d’utiliser les techniques dites de simulation moléculaire, qui permettent de décrire les structures et les mouvements des systèmes biologiques, tels que des protéines interagissant entre elles. Cependant, pour décrire correctement des systèmes de grande taille pour des temps suffisamment long, une grande puissance de calcul est nécessaire.
Proposer des médicaments adaptés pour limiter l'infection des cellules humaines du virus
Des chercheurs du LPCT, coordonnés par Antonio Monari, ont décidé d’utiliser la simulation moléculaire pour comprendre le comportement du Covid et proposer ainsi des médicaments rationnellement développés. Ces travaux se font dans le cadre du projet
"SeekAndDestroy", porté par le LPCT, et réunissant des équipes de Nancy, de Palerme et d’Alcala de Hénares (Madrid). Ces recherches peuvent se faire seulement grâce à l’investissement du CNRS, IDRIS et GENCI qui ont donné un accès prioritaire au
supercalculateur national Jean Zay. En effet, depuis trois semaines plusieurs centaines de processeurs tournent en permanence pour permettre ces calculs, ce qui représente l’équivalent des plusieurs mois de calculs sur une simple machine.
"SeekAndDestroy" : comprendre et détruire
Le projet "SeekAndDestroy" cible deux protéines du virus :
•
la protéine dite « Sars unique domain », qui permet au virus d’échapper aux systèmes de défense de la cellule. Le LPCT a déjà montré que ce mécanisme se fait par le biais de l’interaction avec un brin d’ARN. Perturber cette interaction par des médicaments spécifiques permettrait de
réduire la résistance du virus.
• la protéine « spike » qui permet au virus d’infecter les cellules en interagissant avec la protéine humaine dite ACE2. Les chercheurs du LPCT veulent comprendre les mécanismes de cette interaction pour proposer des médicaments capables de la perturber et donc de limiter, ou empêcher, la capacité du virus d’infecter les cellules humaines.
Grâce aux ressources des calculs mis à disposition et à la force de la collaboration internationale, les chercheurs du LPCT ont déjà obtenus des
résultats importants soumis à la communauté scientifique et restent mobilisés pour comprendre et détruire (
SeekAndDestroy) Covid-19. De plus, ces travaux qui permettent de comprendre les mécanismes moléculaires à la base de l’action, pourront aussi permettre de contrer d’éventuels phénomènes de résistances suite à des mutations du virus.
*
LPCT : Laboratoire de Physique et Chimie Théoriques (CNRS/Université de Lorraine)